|
|
Journal of Advanced Veterinary Research Volume 9, Issue 4, 2019, Pages: 187-196 www.advetresearch.com |
|
|
Evaluation of Some Virulence Factors of Methicillin and Vancomycin Resistant Staphylococcus epidermidis Isolated from Cheese and Human Samples |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Asmaa G. Mubarak1, Mona A. El‐Zamkan2* |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
1Department of Zoonoses, Faculty of Veterinary Medicine, South Valley University, Qena 83523, Egypt. 2Department of Food Hygiene and Control, Faculty of Veterinary Medicine, South Valley University, Qena 83523, Egypt. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Received: 15 September 2019; Accepted: 25 October 2019 (*Corresponding author: m_zam@vet.svu.edu.eg) |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Abstract |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Biofilm formation and enterotoxin production represent major virulence factors of S. epidermidis. Also, biofilm formation is greatly associated with multidrug resistance. So the objective of the present study was to expand the current knowledge regarding the importance and virulence of methicillin and vancomycin resistant S. epidermidis originated from dairy food, food handlers and patients in a hospital, and highlight the possible transmission through foods and food handlers. Biofilm formation was evaluated phenotypically by the tube method and microtiter plate method and genotypically through detection of icaA and icaD genes, while S. epidermidis isolates were investigated for potential enterotoxin production through detection of enterotoxin encoding genes (sea, seb and sec). Among the investigated isolates, phenotypic and genotypic biofilm formation was confirmed in 78.4 and 66.7 % of the isolates, respectively. Regarding enterotoxin encoding genes, it was found that seb gene was the only prevailing gene in the three categories of samples with an incidence of 27.5 %. The findings of this study illustrated the prominent role that played by food handlers in transmission of virulent S. epidermidis to food and subsequently patients. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Keywords: Biofilm; Dairy food; Enterotoxin; Food handlers; S. epidermidis |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Introduction |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Coagulase-negative Staphylococci (CoNS) constitute a major component of the normal human microfloras and have been evaluated as saprophytes. However, over the last 3 decades, the increased number of human infection cases due to CoNS has been documented representing one of the major nosocomial pathogens, with S. epidermidis as the serious significant species (Kloos and Bannerman, 1994; De Silva et al., 2001; Vuong and Otto, 2002; Jean-Baptiste et al., 2011; Becker et al., 2014). An important virulence factor associated with S. epidermidis is its ability to form biofilms and colonize biomaterials (Fey and Olson, 2010). Bacterial ability to form biofilms is of great importance and represents a big challenge not only for the food industry but also for the health facilities, as some strains in their sessile state may endure antimicrobial agents, making the bacterium immensely difficult to be erased (Basanisi et al., 2017). In dairy industry the removal of permanently adhered cells is difficult and imposes the application of strong mechanical force or chemical interruption of the microbial adhesion through using surfactants, sanitizers or heat; even though, the probability that the irreversibly adhered cells will survive even after pasteurization is high (Schlegelova et al., 2008). On the other hand, from the public health hazard point of view, biofilms are contributed to more than 80% of all infections in humans (Kot et al., 2017); S. epidermidis is a major cause of nosocomial and implant-associated infections (Otto, 2009) due to its capacity to adhere to catheters, indwelling medical devices or to colonize different surfaces (Ferreira et al., 2012; Prakash et al., 2016). Biofilm formation in S. epidermidis, is associated with the icaABDC operon presence, which encodes the synthesis of the polysaccharide intercellular adhesion (PIA), which considered an important component in the process of biofilm formation and consists of a b -1,6-linked homoglycan composed of N-acetylglucosamine (Gerke et al., 1998; Mack et al., 2004; Liberto et al., 2009; Oliveira and de Cunha, 2010). The chromosomal intercellular adhesion (ica) locus, consisting of the icaADBC structural; and the icaA and icaD genes have been reported to play a fundamental role in biofilm consistence (Darwish and Asfour, 2013). In the late '50s and early '70s of the twentieth century, a few reports suggested that CoNS might also produce enterotoxins in food poisoning cases (Omori and Kato, 1959; Breckindridge and Bergdoll, 1971). Enterotoxigenic CoNS including S. epidermidis were isolated from food and dairy products (Valle et al., 1990; Marín et al., 1992; Rodríguez et al., 1996; Zell et al., 2008; Even et al., 2010; Rall et al., 2010a) and also from cases of human clinical infections (Da Cuhna et al., 2007; Ataee et al., 2011; Vasconcelos et al., 2011). The presence of sequences homologous to S. aureus enterotoxins has been affirmed in the genomes of CoNS strains used in food processing, associated with human infection and from other environments (Weir et al., 2007; Madhusoodanan et al., 2011). Several studies have shown that some CoNS species including S. epidermidis possess the genes for staphylococcal enterotoxins (SE) and can produce a functional toxin (Becker et al., 2001; Blaiotta et al., 2004; Oliveira et al., 2011). Staphylococcal enterotoxins can be classified into five main groups (sea, seb, sec, sed, and see) (Rojas et al., 2012; De Freitas et al., 2013; Pinheiro et al., 2015). Antibiotic resistance in CoNS, including the clinically-significant species S. epidermidis, represents an important health problem worldwide (Cosgrove, 2006). The prevalence of methicillin-resistant S. epidermidis has been increased widely and glycopeptides particularly vancomycin, which was the recommended treatment has also exhibited a failure in the treatment of some cases as a result of its resistance (Chakraborty et al., 2011; Morgenstern et al., 2016). Biofilms formed by S. epidermidis decrease the metabolic activity of the bacteria and protect it from phagocytosis by effector cells (Schommer et al., 2011; Spiliopoulou et al., 2012; Hanke et al., 2013), also reduced the diffusion of antibiotics that makes it difficult for antibiotics to affect this type of infection (Arciola et al., 2010). Since, there is an association between biofilm production and the antimicrobial resistance in S. epidermidis (Argudín et al., 2015), and many other authors stated that the pathogenicity of S. epidermidis has ascribed to the biofilm formation beside enterotoxins production (Irlinger, 2008; Podkowik et al., 2013; Chaves et al., 2018); so, the intention of our study was to expand the knowledge regarding the virulence potential of methicillin and vancomycin resistant S. epidermidis isolates from dairy foods, food handlers and patients in the hospital through: a) Detection the ability of these strains to phenotypic formation of biofilm; b) Molecular detection of biofilm encoding genes (icaA and icaD) . c) Molecular detection of staphylococcal enterotoxins genes sea, seb, and sec. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Materials and methods |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S. epidermidis isolates A total of 51 S. epidermidis isolates showed resistance to both methicillin and vancomycin antibiotics (MR/VR- S. epidermidis) were obtained in a study conducted by El-Zamkan et al. (2019) at a hospital in Qena City, Egypt, on 210 samples that examined for detection of methicillin and/or vancomycin resistant CoNS including 90 dairy food samples which offered to the patients in the hospital (soft cheese, processed cheese, and yoghurt; 30 samples for each), 60 nasal and hand swabs from food handlers working in the hospital (30 for each) and 60 nasal and diarrheal swabs from patients admitted to the hospital and suffered from diarrhea (30 for each). In this study, the isolates were evaluated for their enterotoxigenecity and ability to form biofilm representing the most important S. epidermidis hazardous virulence factors. Ethical approval is not required in case of food samples while oral consent was obtained from each participant patient. Isolation and detection of methicillin and vancomycin resistant S. epidermidis Human samples were collected in sterile plastic containers with sterile saline solution (0.9%NaCl) by using sterile swabs and transported to the laboratory, then one ml of each human sample was inoculated in 9 ml sterile buffered peptone water (Oxoid CM0509, Basingstoke, Hampshire, England), while 10 gm of each food sample was homogenized in 90 ml sterile buffered peptone water (Oxoid, CM0509). Buffered peptone water was incubated overnight at 37°C. Then, a loopful of each sample was streaked on Mannitol Salt Agar (Merck, Germany) and incubated aerobically at 37°C for 24 h for growth; pink colonies were considered as CoNS. Identification of CoNS species was done according to Kloos and Schleifer, (1975) and Bannerman, (2003). Resistance to methicillin and vancomycin was detected phenotypically according to the guidelines of the National Reference Centre for Antimicrobial Susceptibility and internationally recognized standards of the Clinical and Laboratory Standards Institute (CLSI, 2014) on Muller Hinton agar (Merck, Germany) using the diffusion disk method. Later, the isolates that showed phenotypic antibiotic resistance were submitted to PCR to confirm presence of mecA gene F (5'- GTA GAA ATG ACT GAA CGT CCG ATA A-3') and R (5'-CCA ATT CCA CAT TGT TTC GGT CTA A-3') (McClure et al., 2006), and vanA gene F (5'- GGGAAAACG ACAATTGC -3') and R (5'- GTACAATGCGGCCGTTA -3') (Depardieu et al., 2004). Biofilm Formation tests Tube method (TM) Biofilm production was examined by the tube adherence test proposed by Christensen et al. (1982) in which 10 mL of Trypticase soy broth with 1% glucose was inoculated with a loopfull of the overnight culture on nutrient agar individually. Broths were incubated at 37 °C for 24 h. The cultures were decanted and tubes were washed with phosphate buffer saline (pH 7.3). The tubes were dried and stained with 0.1% crystal violet. Excess stain was washed with deionized water. Tubes were dried in an inverted position and observed for biofilm formation. Biofilm Production was considered positive when a visible film lined the wall and bottom of the tube. Ring formation at the liquid interface was not indicative of biofilm formation. Microtiter plate (MTP) method The technique was done as described by Stepanović et al. (2000). Each MR/VR- S. epidermidis isolate was grown on Tryptic-Soy Agar with 1% glucose at 37 °C overnight, as the growth medium. Then a dilution 1:100 was prepared in Tryptic soy broth (TSB) medium supplemented with glucose (1%) followed by filling three wells of a sterile 96- well flat-bottomed polystyrene tissue culture plate with a lid with 200µL of aliquots of the diluted culture; negative control wells contained uninoculated sterile broth. The plates were covered and incubated for 24 h at 37 °C. The contents of each well were gently aspirated, and the wells were washed three times with 250 µL of phosphate-buffered saline (PBS). The remaining adhered bacterial cells were fixed with 200µL of 99% methanol for 15 min., then plates were air-dried after methanol removal, After that wells were stained with 0.2 mL of 0.2% crystal violet (CV) for five min. Then, wells were rinsed twice with distilled water and dried thoroughly. For the quantification of biofilm growth, the adherent cells were resolubilized with glacial acetic acid (33%). The OD of the resulting solutions in each well was measured at 570 nm with a microplate reader. Based on the OD of the bacterial film, all strains were classified into the following categories: “OD≤ODc: non–adherent, ODc<OD≤2×ODc: weakly adherent, 2×ODc<OD≤4×ODc: moderately adherent, 4×ODc<OD: strongly adherent”. All tests were carried out in triplicates and the results were averaged. Isolates were considered biofilm-positive when they have an OD 570 nm > 0.2. Each isolate was tested in triplicate and the results were averaged. Molecular detection of enterotoxins and biofilm associated genes Biofilm encoding genes and enterotoxin genes detected in this study and their amplification conditions are listed in Table 1. Primers were supplied from Metabion (Germany). DNA extraction DNA extraction was performed using the QIAamp DNA Mini kit (Qiagen, Germany, GmbH) with modifications from the manufacturer’s recommendations. Briefly, 200 µL of the sample suspension was incubated with 10 µL of proteinase K and 200 µL of lysis buffer at 56 °C for 10 min. After incubation, 200 µL of 100% ethanol was added to the lysate. The sample was then washed and centrifuged following the manufacturer’s recommendations. Nucleic acid was eluted with 100 µL of elution buffer provided in the kit. Biofilm production and analysis of icaA/D genes Primers were utilized in a 25 µL reaction containing 12.5 µL of Emerald Amp Max PCR Master Mix (Takara, Japan), one µL of each primer of 20 pmol concentrations, 4.5 µL of water, and 6 µL of DNA template. The reaction was performed in an Applied Biosystem 2720 thermal cycler. Enterotoxins detection Primers of sea, seb, and sec genes were utilized in a 50 µL reaction containing 25 µL of EmeraldAmp Max PCR Master Mix (Takara, Japan), one µL of each primer of 20 pmol concentration, 8 µL of water, and 7 µL of DNA template. The reaction was performed in an Applied Biosystem 2720 thermal cycler. Analysis of the PCR Products The products of PCR were separated by electrophoresis on 1.5% agarose gel (Applichem, Germany, GmbH) in 1x TBE buffer at room temperature using gradients of 5V/cm. For gel analysis, 20 µL of the uniplex PCR products and 40 µL of the multiplex PCR products were loaded in each gel slot. Gelpilot 100 bp and 100 bp plus DNA ladders (Qiagen, Germany, GmbH) were used to determine the fragment sizes. The gel was photographed by a gel documentation system (Alpha Innotech, Biometra) and the data was analyzed through computer software. Statistical analysis Bacterial count and OD variables, significant factors (P≤0.05) and agglomerative hierarchical clustering (ACH) performed using XLSTAT 2016 software for Microsoft Excel® (Microsoft®, WA, USA). |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Results |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Of the all 51 methicillin and vancomycin resistant S. epidermidis isolates obtained from cheese, food handlers and patients in a hospital in Qena Governorate, 23.5, 62.7 and 19.6% were positive for the presence of icaA, icaD and both icaA & icaD genes (Table 2 and Figs. 1 and 2). Out of the 51 isolates, 27 versus 40 isolates showed biofilm-forming phenotypes using tube and microtiter plate (MPT) methods, respectively; and the later ranged from weak to strong adherent (Table 4). The frequency distribution of genes and biofilm phenotype detected is shown in Fig. 3. There were no statistically significant differences between the presence of icaA and/or icaD gene and biofilm formation using MTP method (P> 0.05), while the accompanying presence of both icaA and icaD was significantly associated with biofilm phenotype (P< 0.05). Table 1. Primers sequences, target genes, amplicon sizes and cycling conditions.
Table 2. Prevalence of icaA and icaD genes in S. epidermidis isolates and phenotypic biofilm production evaluated by microtitre plate method (MPT) and tube method.
a=strong adherent; b= moderate adherent; c= weakly adherent Table 3. Prevalence of enterotoxin genes in S. epidermidis isolates
Table 4. Virulence profile of S. epidermidis strains isolated from cheese, food handlers and hospitalized patients.
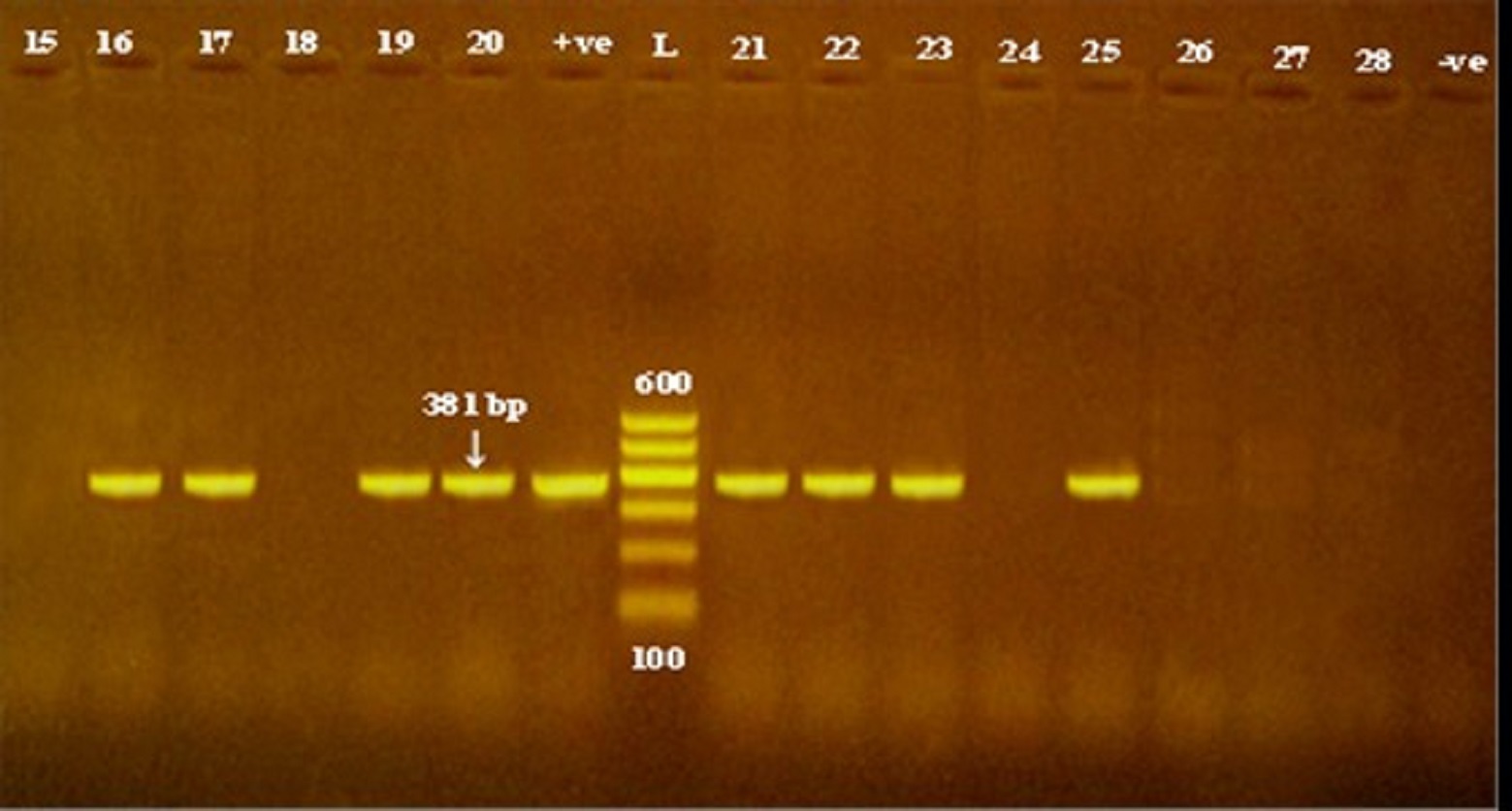
According to data showed in Table 3, of all the investigated isolates 27.5 % (14/51) were harboring seb gene, including 30.8% (4/13) of each dairy food and food handlers’ isolates and 24% (6/25) of hospitalized patients, while sea gene was found in only one S. epidermidis isolate originated from cheese sample (Fig. 4).
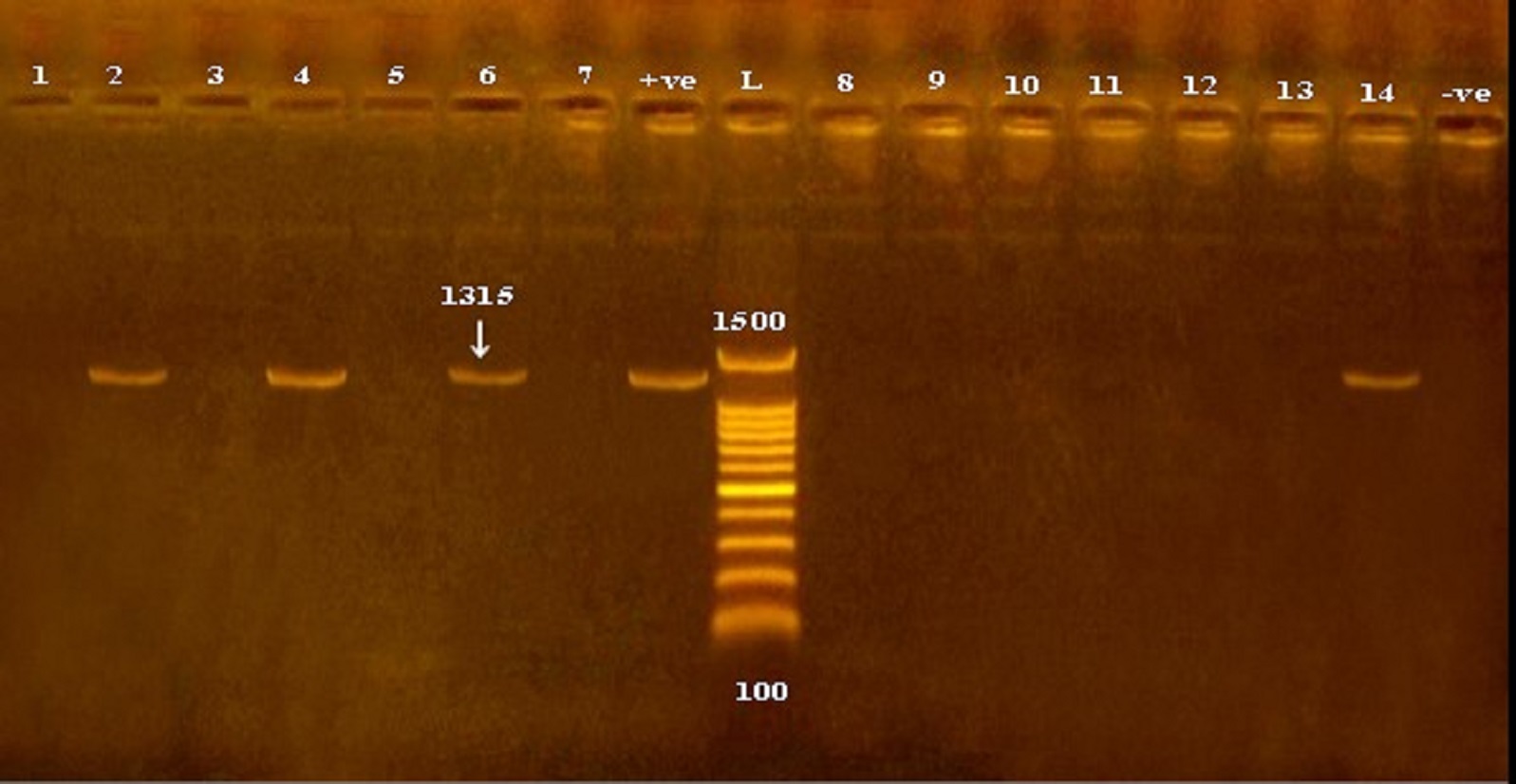
Fig. 1. Detection of icaA gene in methicillin and vancomycin resistant S. epidermidis isolates obtained from cheese and human samples. L: Ladder, +ve: positive control, -ve: negative control.
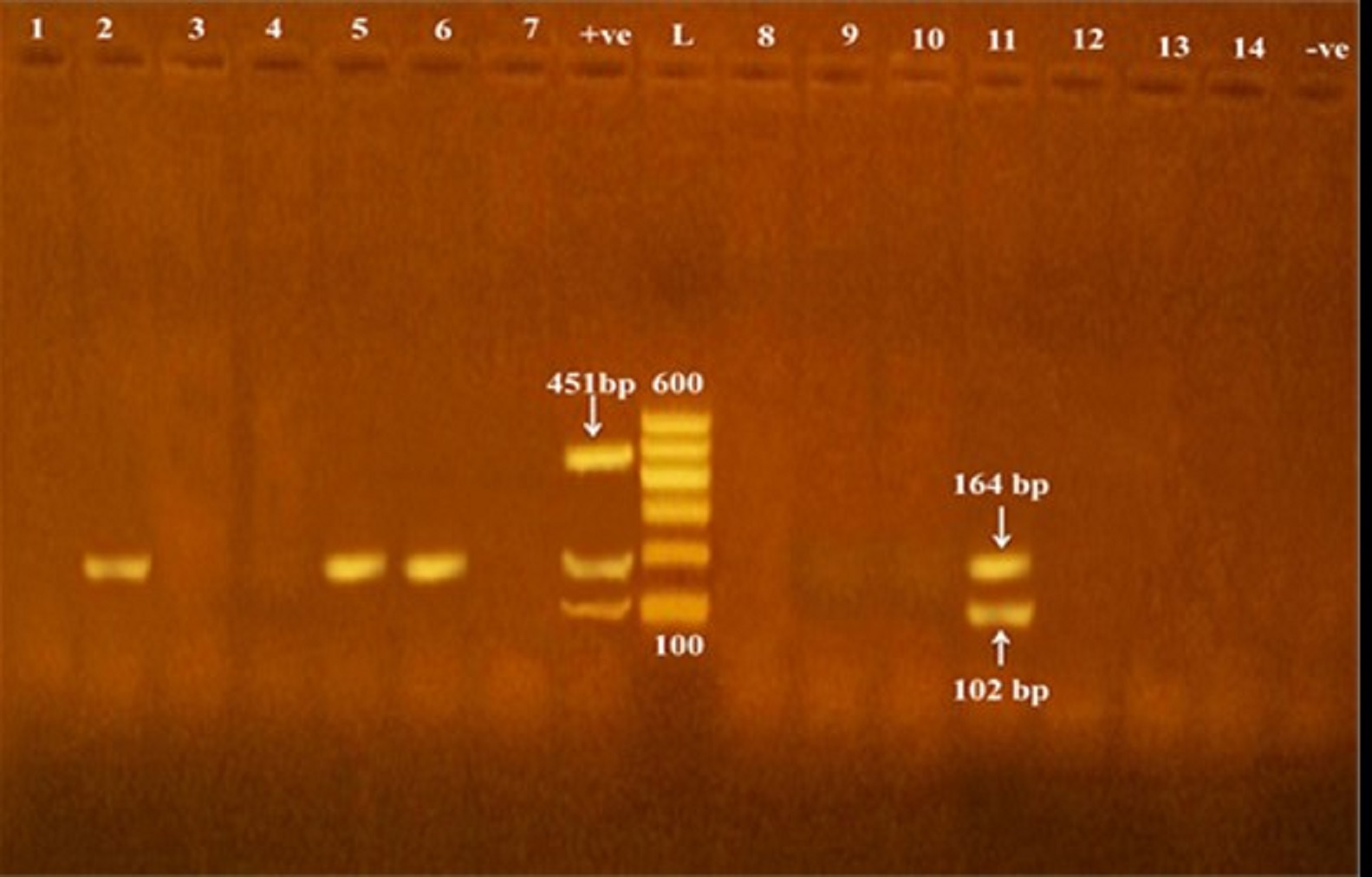
Fig. 2. Detection of icaD gene in methicillin and vancomycin resistant S. epidermidis isolates obtained from cheese and human samples. L: Ladder, +ve: positive control, -ve: negative control. Fig. 3. Frequency distribution of biofilm genes in relation to phenotypic biofilm formation (*SA=strong adherent; MA= moderate adherent; WA= weakly adherent; icaD*=icaD gene only; icaA= icaA gene only).
Fig. 4. Detection of sea, seb and sec genes encoding staphylococcal enterotoxin production in methicillin and vancomycin resistant S. epidermidis isolates obtained from cheese and human samples. L: Ladder, +ve: positive control, -ve: negative control. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Fig. 5. Dendrogram showing the relatedness between MR/VR S. epidermidis isolates. The dendrogam is based on the presence/absence of seb and biofilm-forming genes (icaA and icaD). |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Discussion |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
There is a limited data regarding phenotypic and genotypic biofilm production patterns in S. epidermidis isolated from dairy food. Additionally, the incidences of strains that possessed icaA and/or icaD genes and showed a phenotypic biofilm formation in the current work should be interpreted with caution because the study was confined to a certain group of small S. epidermidis isolates number which all shared resistance to both methicillin and vancomycin which may be the reason for reporting such higher results in cheese than Schlegelová et al. (2008), Chajęcka-Wierzchowska et al. (2019), and in food handlers than Udo et al. (2009), while in patients it was higher than Ninin et al. (2006); Gad et al. (2009); Saising et al. (2012); Ebrahimi et al. (2014) and Kord et al. (2018). However, in human samples, many other authors like Smith et al. (2008) and Piechota et al. (2018) recorded higher results as S. epidermidis is more abundant in human including patients and healthy persons (Schlegelova et al., 2008; Jaglic et al. 2010). From the presented results, it is clear that icaD gene was the most detected biofilm gene in the isolates either separately or in combination with icaA. In the same way, de Mello et al. (2014) and Chajęcka-Wierzchowska et al. (2019) detected icaD gene in a higher proportion than icaA gene S. epidermidis isolated from cheese and food handlers, respectively. Unlike our results, icaA was the most detected gene in S. epidermidis isolated by Pinheiro et al. (2016) and by Hasanvand et al. (2019), while no mentioned difference between the incidence of icaA and icaD genes in S. epidermidis collected from milk samples was found by Abbondio et al. (2019). De Silva et al. (2002) described an association between the ica operon and quantitative biofilm formation. In the current study, the emphasis is on the importance of biofilm formation in the pathogenesis of MR/VR- S. epidermidis, particularly the role that may be played by icaA and/or icaD genes in the phenotype of the formed biofilm. In the current study, most isolates contained icaD gene produced biofilm ranged from weak to strong; while the concomitant presence of both icaA and icaD was significantly associated with biofilm phenotype (P< 0.05) as all strains that co-expressed icaA with icaD were strong biofilm formers. Similarly, Cafiso et al. (2004); Pinheiro et al. (2016) and Chajęcka-Wierzchowska et al. (2019) found that the presence of icaA and icaD was significantly associated with the biofilm formation results. Unlike these findings, Ninin et al. (2006) could not find such a relation. Also, it was found that only two isolates obtained from nasal swabs of patients were found to have only icaA gene and theses isolates associated with weak biofilm phenotype. This genotypic, phenotypic correlation was explained in a study conducted by Gerke et al. (1998) where they found that presence of icaA alone revealed a weak N-acetylglucosaminyl transferase activity, while its full activity is only achieved when icaD is co-transcribed with icaA; the PIA consists of a b-1,6-linked homoglycan composed of N-acetylglucosamine, which is a substrate for the glycosyltransferase reaction in PIA biosynthesis. Results illustrated in this study revealed that, neither icaA nor icaD could be detected in non-biofilm formers which originated from cheese and food handlers and this disagree with Los et al. (2010); Abu Taleb et al. (2012); Abbondio et al. (2019); Chajęcka-Wierzchowska et al. (2019). On the other hand, in this study six isolates obtained from nasal swab of patients as well as isolates obtained by Bartoszewicz et al. (2004); Qin et al. (2007); Schommer et al. (2011); Darwish and Asfour (2013); Lira et al. (2016) and Pinheiro et al. (2016) showed the ability to produce biofilm while they were ica negative strains; contrarily, Rohde et al. (2004) and Salgueiro et al. (2017) reported that PCR positivity for icaA/icaD genes, can also be found in non-biofilm producers. This conflict between studies could be contributed to many theories a): Biofilm formation is largely a strain-specific trait (Mack et al., 2004); b): Regulation of the ica operon appears to be very complex. Production of PIA is certainly subjected to on-off switching and may be involved in S. epidermidis phase-variation that might improve bacterial survival and growth under changing environmental conditions in vivo (Ninin et al., 2006). The microtitre plate method is considered as the golden standard method for biofilm detection as it quantified the formed biofilm in 40/51 S. epidermidis, while tube method identified 24/51 isolates as a non-biofilm producer which may be due to the difficulty to differentiate between weak and non-biofilm because the interpretation of the results of tube method varies according to each person observation, also differences in biofilm formation between phenotypic methods may be due to the effect of different culture media, pH, temperature, and osmotic pressure (Deka, 2014). The high incidences of phenotypic and genotypic biofilm S. epidermidis producer in this study as well as other studies performed by De Silva et al. (2002); Cafiso et al. (2004); Prasad et al. (2012); Sahal and Bilkay (2014); Argudín et al. (2015) and Chajęcka-Wierzchowska et al. (2019) confirmed that biofilm formation and antibiotic resistance are considered to be one of the main virulence factors and are associated with each other and are regarded as important markers that differentiate commensal and pathogenic strains; also strains that are stronger biofilm formers are often multidrug resistant. Although CoNS safety hazards associated with the presence of methicillin and vancomycin antibiotic resistance and the mutual role that played by food and food handlers in its transmission have been illustrated in the latter study performed by El-Zamkan et al. (2019) ‘unpublished data’, it was important to investigate some CoNS isolates (S. epidermidis) found in that study for their positivity for enterotoxin genes, which have been reported for its existence and for being frequent causes of food poisoning outbreaks and that initiated through consumption dairy food (Rosec et al., 1997; Carmo et al., 2002; Veras et al., 2003; Park et al., 2011). The molecular method can be considered as the routine analytical method for SE detection. Although, staphylococcal enterotoxin SEA is the most common cause of staphylococcal food poisoning worldwide, but the involvement of other staphylococcal enterotoxins has been also demonstrated (Argudín et al., 2010). The results of this study revealed that seb gene was the most prevailing gene (30.8%) in S. epidermidis isolates originated from cheese samples followed by sea gene (7.7%), while sec gene could not be located in these isolates. Results obtained by Even et al. (2010) pointed out that 76% of the strains carrying genes encoding staphylococcal toxin belonged to S. epidermidis species isolated from fermented food including cheese, while, Guimarães et al., (2013) found that 63.2% S. epidermidis strains isolated from milk showed enterotoxin-encoding genes and sea and seb were the most dominating, while, Rodrigues et al. (2017) detected enterotoxin genes in S. epidermidis isolated from 3 different processing plants of fresh cheese but none of sea, seb or sec was amongst them. Contrarily, Rall et al. (2010a), Siqueira et al. (2017) and Abbondio et al. (2019) reported that none of the enterotoxin genes (sea, seb, sec) could be detected in S. epidermidis isolated from milk. Also, the results of this study revealed that seb gene only could be detected in 30.8 and 24% of S. epidermidis isolates obtained from food handlers and patients, respectively. This obtained result comes in parallel to data recorded by Ahanotu et al. (2006) and Sospedra et al. (2012) which proved that seb gene is the most common gene in coagulase negative Staphylococci identified from foods and food handlers. The gene was detected in closely related result in multidrug resistant S. epidermidis isolated from food handlers in a hospital as 35% by Francisco and Moreno (2018); also in a study achieved by Udo et al. (2009) 38% of S. epidermidis isolates produced different SEs with the majority for seb gene. In contrary to this result, a lower incidence of seb gene (17.2%) could be detected by Rall et al. (2010b). The enterotoxin production by S. epidermidis was also studied by several authors as Rapini et al. (2005) who observed that S. epidermidis isolated from food handlers produced sea and sed in 96.4%. A smaller prevalence of CoNS enterotoxin-encoding genes was observed by Crass and Bergdoll (2007), as 16.5% of CoNS strains obtained from food handlers. While the higher prevalence of seb gene producing S. epidermidis strains isolated from food handlers was observed by Santos (2003) as 63.2%. On the other hand, Çepoğlu et al., (2010) couldn’t detect seb gene in S. epidermidis isolates from food handlers and detected only see gene. Regarding hospitalized patients, closely related results to ours were obtained by Pinheiro et al. (2015) who recorded that 30% of S. epidermidis isolates obtained from patients contained seb gene. The production of the classical enterotoxins sea, seb, and sec by clinical isolates of multidrug resistant S. epidermidis from hospitalized patients has been also described by Cunha et al. (2007); Barretti et al. (2009); Rojas et al. (2012) and Vasconcelos et al. (2011). The differences between studies may be related to variation in the isolates studied, including the number, nature and geographic origin of the strains. Clustering the isolates according to its detected virulence factors (The presence or absence of icaA, icaD and seb genes) illuminated the close relationship between samples of human handlers and dairy foods followed by their closer relationship with diarrheal samples, while the nasal swabs of patients were located in a separate arm (Fig. 5). This cluster highlights the mutual role played by food handlers in transmitting virulent S. epidermidis to food through cross contamination and the role of these strains in initiating foodborne diseases through food consumption. This was also observed by Doulgeraki et al. (2017) who stated that methicillin resistant Staphylococci could produce biofilms by contamination from human handlers rather than from food itself; yet in this study food handlers cannot be considered as standard community isolates as the hospital personnel can act as a reservoir or as a vector for clinical S. epidermidis strains (Milisavljevic et al., 2005). |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Conclusion |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
The data obtained in this study pointed out the linkage between the antibiotic resistance and biofilm formation by S. epidermidis and also the relation of icaA and icaD genes expression and biofilm phenotype. Additionally, the occurrence of staphylococcal enterotoxin genes in isolates of this study highlights their potentiality to cause food poisoning. Hence, Biofilm together with enterotoxin production are the most important virulence factors of S. epidermidis. All the above findings are dominated by the conspicuous role played by food handlers in the transmission of the virulent S. epidermidis to food or patients. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Acknowledgments |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
The authors acknowledge the contributions from the members of the hospital in collection of the samples. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Conflict of Interests |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
The authors declare that they have no conflict of interest. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
References |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Abbondio, M., Fois, I., Longheu, C., Azara, E., Tola, S., 2019. Biofilm production, quorum sensing system and analysis of virulence factors of Staphylococcus epidermidis collected from sheep milk samples. Small Ruminant Research 174, 83-87. Abu Taleb, A.M.F., Mohamed, M.S., Abdel-Latif, R.S., Gouda, M., 2012. The role of ica operon and biofilm formation in coagulase negative staphylococcal infection. Egyptian J. Med. Microbiol 21, 21–32. Ahanotu, E., Alvelo-Ceron, D., Ravita, T., Gaunt, E., 2006. Staphylococcal enterotoxin B as a biological weapon: recognition, management, and surveillance of Staphylococcal enterotoxin. Appl. Biosaf. 11, 120-126. Arciola, C.R., Baldassarri, L., Montanaro, L., 2010. In catheter infections by Staphylococcus epidermidis the intercellular adhesion (ica) locus is a molecular marker of virulent slime-producing strains. J. Biomed. Mater. Res. 59, 557-562. Argudín, M.Á., Mendoza, M.C., Rodicio, M.R., 2010. Food poisoning and Staphylococcus aureus enterotoxins. Toxins 2, 1751– 1773. Argudín, M.A., Vanderhaeghen, W., Vandendriessche, S., Vandecandelaere, I., Denis, O., Coenye, T., Butaye, P., 2015. Biofilm formation of ica operon-positive Staphylococcus epidermidis from different sources. APMIS Journal of Pathology, Microbiology and Immunology 123, 1081-1089. Ataee, R.A., Mehrabi-Tavana, A., Izadi, M., Hosseini, S.M., Ataee, M.H., 2011. Bacterial meningitis: a new risk factor. Journal of Medical Sciences Research 16, 207–210. Bannerman, T.L., 2003. Staphylococcus, Micrococcus, and other catalase-positive cocci that grow aerobically. In: Manual of Clinical Microbiology American Society Microbiology. PR Murray, Baron EJ, Jorgensen JH, Pfaller MA, Yolken RH (eds), Washington, pp. 384-404. Barretti, P., Montelli, A.C., Batalha, J.E., Caramori, J.C., Cunha, M.L., 2009. The role of virulence factors in the outcome of staphylococcal peritonitis in CAPD patients. BMC Infect. Dis. 9, 212-219. Bartoszewicz, M., Nowicka, J., Janikowska, E., Przondo-Mordarska, A., 2004. Adhesion dependence of coagulase-negative staphylococci to biomaterials and PIA polysacchapjde synthesis on icaADBC operon presence. Med. Dosw. Mikrobiol. 56, 225–230. Basanisi, M.G., La Bella, G., Nobili, G., Franconieri, I., La Salandra, G., 2017. Genotyping of methicillin-resistant Staphylococcus aureus (MRSA) isolated from milk and dairy products in South Italy. Food Microbiology 62, 141-146. Becker, K., Heilmann, C., Peters, G., 2014. Coagulase-negative Staphylococci. Clinical Microbiology Reviews 27, 870–926. Becker, K., Keller, B., von Eiff, C., Bruck, M., Lubritz, G., Etienne, J., 2001. Enterotoxigenic potential of Staphylococcus intermedius. Appl. Environ. Microbiol. 67, 5551-5557. Blaiotta, G., Ercolini, D., Pennacchia, C., Fusco V., Casaburi, A, Pepe, O, Villani, F., 2004. PCR detection of staphylococcal enterotoxin genes in Staphylococcus spp strains isolated from meat and dairy products. Evidence for new variants of seG and seI in S. aureus AB-8802. J. Appl. Microbiol. 97, 719-30. Breckindridge, J.C., Bergdoll, M.S., 1971. Outbreak of food borne gastroenteritis due to a coagulase-negative enterotoxin producing Staphylococcus. The New England Journal of Medicine 284, 541–543. Cafiso, V., Bertuccio, T., Santagati, M., Campanile, F., Amicosante, G., Perilli, M.G., Selan, L., Artini, M., Nicoletti, G., Stefani, S., 2004. Presence of the ica operon in clinical isolates of Staphylococcus epidermidis and its role in biofilm production. Clinical Microbiology and Infection 10, 1081-1088. Carmo, L.S., Dias, R.S., Linardi, V.R., Sena, M.J., Santos, D.A., Faria, M.E., Pena, E.C., Jett, M., Heneine, L.G., 2002. Food poisoning due to enterotoxigenic strains of Staphylococcus present in Minas cheese and raw milk in Brasil. Food Microbiology 19, 9–14 Çepoğlu, H., Vatansever, L., Oral, N.B., 2010. Isolation of Staphylococci from Food Handlers and Investigation of Their Enterotoxigenicity and Susceptibility to Some Antibiotics Kafkas Univ. Vet. Fak Derg Research Article 16 (Suppl-A): S1-S5. Chajęcka-Wierzchowska, W., Zadernowska, A., Gajewska, J., 2019. S. epidermidis strains from artisanal cheese made from unpasteurized milk in Poland - Genetic characterization of antimicrobial resistance and virulence determinants. International Journal of Food Microbiology 294, 55-59. Chakraborty, S., Mahapatra, M., Bal, M., Roy, S., 2011. Isolation and identification of vancomycin resistant Staphylococcus aureus from post-operative pus sample. Al Amen J. Med. Sci. 2, 152–168. Chaves, R.D., Pradella, F., Turatti, M.A., Amaro, E.C., da Silva, A.R., Farias, A.S., Pereira, J.L., Christensen, G.D., Bisno, A.L., Simpsom, W.A., Beachey, E.H., 1982. Adherence of slime producing strains of Staphylococcus epidermidis to smooth surfaces. Infectious Immunity 37, 318–326. Ciftci, A., Findik, A., Onuk, A., Savasan, S., 2009. Detection of methicillin resistance and slime factor production of staphylococcus aureus in bovine mastitis. Brazilian Journal of Microbiology 40, 254-261. Clinical and Laboratory Standards Institute (CLSI), 2014. Performance standards for susceptibility testing, 24 h informational supplement, vol. 34: Wayne, PA: National Committee for Clinical Laboratory Standards. Cosgrove, S.E., 2006. The relationship between antimicrobial resistance and patient outcomes: mortality, length of hospital stay, and health care costs. Clinical Infectious Diseases 15, S82-S89. Crass, B., Bergdoll, M.S., 2007. Involvement of coagulase-negative staphylococci in toxic shock syndrome. Journal of Clinical Microbiology 23, 43–45. Cunha, M.L.R.S., Calsolari, R.A.O., Araújo-Júnior, J.P., 2007. Detection of enterotoxin and toxic shock syndrome toxin 1 genes in Staphylococcus, with emphasis on coagulase-negative staphylococci Microbiol. Immunol. 51, 381–390. Da Cuhna, M., Calsolari, R.A., Júnior, J.P., 2007. Detection of enterotoxin and toxic shock syndrome toxin 1 genes in Staphylococcus, with emphasis on coagulase-negative Staphylococci. Microbiology and Immunology 51, 381–390. Darwish, S.F., Asfour, H.A., 2013. Investigation of biofilm forming ability in Staphylococci causing bovine mastitis using phenotypic and genotypic assays. The Scientific World Journal 2013, Article ID 378492, 9 pages. De Freitas, G.F., Nóbrega, D.B., Richini-Pereira, V.B., Marson, P.M., de Figueiredo, J.C., Langoni, H., 2013. Enterotoxin genes in coagulase-negative and coagulase-positive staphylococci isolated from bovine milk. J. Dairy Sci. 96, 2866–2872. de Mello, J.F., da Rocha, L.B., Lopes, L.S., Frazzon, J., da Costa, M., 2014. Sanitary quality, occurrence and identification of Staphylococcus spp. in food services. Braz. J. Microbiol. 45, 211-216. De Silva, G.D.I., Justice, A., Wilkinson, A.R., Buttery, J., Herbert, M., Day, N.P.J., Peacock, S.J., 2001. Genetic population structure of coagulase-negative staphylococci associated with carriage and disease in preterm infants. Clin. Infect. Dis. 33, 1520-1528. De Silva, G.D.I., Kantzanou, M., Justice, A., Massey, R.C., Wilkinson, A.R., Day, N.P.J., Peacock, S.J., 2002. The ica-operon and biofilm production in coagulase-negative Staphylococci associated with carriage and disease in a neonatal intensive care unit. Journal of Clinical Microbiology 40, 382-388. Deka, N., 2014. Comparison of tissue culture plate method, tube method and Congo red agar method for the detection of biofilm formation by coagulase negative Staphylococcus isolated from non-clinical isolates. Int. J. Curr. Microbiol. App. Sci. 3, 810-815. Depardieu, F., Perichon, B., Courvalin, P., 2004. Detection of the van Alphabet and Identification of Enterococci and Staphylococci at the Species Level by Multiplex PCR. J. Clin. Microbiol. 42, 5857–5860. Doulgeraki, A.I., DiCiccio, P., Ianieri, A., Nychas, G.J.E., 2017. Methicillin resistant food-related Staphylococcus aureus: a review of current knowledge and biofilm formation for future studies and applications. Res. Microbiol. 168, 1-15. Ebrahimi, A., Ghasemi, M., Ghasemi, B., 2014. Some virulence factors of staphylococci isolated from wound and skin infections in Shahrekord, IR Iran. Jundishapur J. Microbiol. 7, e9225. El-Zamkan, M.A., Mubarak, A.G., Ali, A.O., 2019. Prevalence and phylogenetic relationship among methicillin and vancomycin resistant Staphylococci isolated from hospital’s dairy food, food handlers, and patients. Journal of Advanced Veterinary and Animal Research. 6, 463-471. Even, S., Leroy, S., Chalier, C., Zakour, N. B., Chacornac, J., Lebert, I., Jamet, E., Desmonts, M., Coton, E., Pochets, S., Donnio, P. Gautier, M., Talon, R. Le Loir, Y., 2010. Low occurrence of safety hazards in coagulase negative Staphylococci isolated from fermented foodstuffs. Int. J. Food Microbiol. 139, 87–95. Ferreira, F.A., Souza, R.R., Bonelli, R.R., Américo, M.A., Fracalanzza, S.E.L., Figueiredo, A.M.S., 2012. Comparison of invitro and invivo systems to study ica-independent Staphylococcus aureus biofilms. J. Microbiol. Meth. 88, 393-398. Fey P.D., Olson, M.E., 2010. Current concepts in biofilm formation of Staphylococcus epidermidis. Future Microbiology 5, 917–933. Francisco, J.J., Moreno, B., 2018. Importance of food handlers as a source of enterotoxigenic coagulase negative staphylococci. Zentralbl Bakteriol Mikrobiol Hygiene 181, 464-474. Gad, G.F., El-Feky, M.A., El-Rehewy, M.S., Hassan, M.A., Abolella, H., El-Baky, R.M., 2009. Detection of icaA, icaD genes and biofilm production by Staphylococcus aureus and Staphylococcus epidermidis isolated from urinary tract catheterized patients. J. Infect. Dev. Ctries. 3, 342–351. Gerke, C., Kraft, A., Süßmuth, A., Schweitzer, O., Götz, F., 1998. Characterization of the N-Acetylglucosaminyltransferase activity involved in the biosynthesis of the Staphylococcus epidermidis Polysaccharide Intercellular Adhesin. J. Biol. Chem. 273, 18586–18593. Guimarães, F. F., Nóbrega, D.B., Richini-Pereira, V.B., Marson, P.M., Pantoja, J.C.F., Langoni, H., 2013. Enterotoxin genes in coagulase-negative and coagulase-positive Staphylococci isolated from bovine milk. J. Dairy Sci. 96, 2866-2872. Hanke, M.L., Heim, C.E., Angle, A., Sanderson S. D., Kielian T., 2013. Targeting macrophage activation for the prevention and treatment of Staphylococcus aureus biofilm infections. J. Immunol. 190, 2159–2168. Hasanvand, H., Teymouri, F., Ohadi, E., Azadegan, A., Kalani, B.S., 2019. Biofilm Formation in Staphylococcus epidermidis Isolated from Hospitalized Patients. Arch. Clin. Infect. Dis. 14, e64496. Irlinger, F., 2008. Safety assessment of dairy microorganisms: Coagulase-negative Staphylococci. Int. J. of Food Microbiol. 126, 302–310. Jaglic, Z., Michu, E., Holasova, M., Vlkova, H., Babak, V., Kolar, M., Bardon, J., Schlegelova, J., 2010. Epidemiology and characterization of Staphylococcus epidermidis isolates from humans, raw bovine milk and a dairy plant. Epidemiol. Infect. 138, 772–782. Jean-Baptiste, N., Benjamin, D., Cohen-Wolkowiez, M., Fowler, V.J., Laughon, M., Clark, R.H., Smith, P.B., 2011. Coagulase-negative Staphylococcal infections in the neonatal intensive care unit. Infect. Control. Hosp. Epidemiol. 32, 679–686. Kloos, W.E., Bannerman, T.L., 1994. Update on clinical significance of coagulase-negative Staphylococci. Clin. Microbiol. Rev. 7, 117-140. Kloos, W.E., Schleifer, K.H., 1975. Simplified scheme for routine identification of human Staphylococcus species. J. Clin. Microbiol. 1, 82–88. Kord, M., Ardebili, A., Jamalan, M., Jahanbakhsh, R., Behnampour, N., Ghaemi, E.A., 2018. Evaluation of Biofilm Formation and Presence of ica Genes in Staphylococcus epidermidis Clinical Isolates. Osong Public Health Res. Perspect. 9, 160–166. Kot, B., Wierzchowska, K., Grużewska, A., Lohinau, D., 2017. The effects of selected phytochemicals on biofilm formed by five methicillin-resistant Staphylococcus aureus. Natural Product Research 32, 1299–1302. Liberto, M.C., Matera, G., Quirino, A., 2009. Phenotypic and genotypic evaluation of slime production by conventional and molecular microbiological techniques. Microbiol. Res. 164, 522–528. Lira, M.C., Givisiez, P.E.N., de Sousa, F.G.C., Magnani, M., de Souza E.L., Spricigo, D.A., Gebreyes, W.A., de Oliveira, J.P., 2016. Biofilm-forming and antimicrobial resistance traits of Staphylococci isolated from goat dairy plants. J. Infect. Dev. Ctries. 10, 932-938. Los, R., Sawicki, R., Juda, M., 2010. A comparative analysis of phenotypic and genotypic methods for the determination of the biofilm-forming abilities of Staphylococcus epidermidis. FEMS Microbiol. Lett. 10, 97–103. Mack, D., Becker, P., Chatterjee, I., Dobinsky, S., Knobloch, J.K., Peters, G., Rohde, H., Herrmann, M., 2004. Mechanisms of biofilm formation in Staphylococcus epidermidis and Staphylococcus aureus: functional molecules, regulatory circuits, and adaptive responses. Int. J. Med. Microbiol. 294, 203–212. Madhusoodanan, J., Seo, K.S., Remortel, B., Park, J.Y., Hwang, S.Y., Fox, L.K., Park, Y.H., Deobald, C.F., Wang, D., Liu, S., Daugherty, S.C., Gill, A.L., Bohach, G.A., Gill, S.R., 2011. An enterotoxin-bearing pathogenicity island in Staphylococcus epidermidis. J. Bacteriol. 193, 1854–1862 Marín, M.E., de la Rosa, M.C., Cornejo, I., 1992. Enterotoxigenicity of Staphylococcus strains isolated from Spanish dry-cured hams. Appl. Environ. Microbiol. 58, 1067-1069. McClure, J.A., Conly, J.M., Lau, V., Elsayed, S., Louie, T., Hutchins, W., Zhang, K., 2006. Novel multiplex PCR assay for detection of the staphylococcal virulence marker Panton-Valentine leukocidin genes and simultaneous discrimination of methicillin-susceptible from resistant staphylococci. J. Clin. Microbiol. 44, 1141-1146. Mehrotra, M., Wang, G., Johnson, W.M., 2000. Multiplex PCR for Detection of Genes for Staphylococcus aureus Enterotoxins, Exfoliative Toxins, Toxic Shock Syndrome Toxin 1, and Methicillin Resistance. Journal of Clinical Microbiology 38, 214-219. Milisavljevic, V., Wu, F., Cimmotti, J., Haas, J., Della-Latta, P., Larson, E., Saiman, L., 2005. Genetic relatedness of Staphylococcus epidermidis from infected infants and staff in the neonatal intensive care unit. Am. J. Infect. Control 33,341-347. Morgenstern, M., Erichsen, C., von Ruden, C., Metsemakers, W.J., Kates, S.L., Moriarty, T.F., 2016. Staphylococcal orthopaedic device-related infections in older patients. Injury 47, 1427–1434. Ninin, E., Caroff, N., Espaze, E., 2006. Assessment of ica operon carriage and biofilm production in Staphylococcus epidermidis isolates causing bacteraemia in bone marrow transplant recipients. Clin. Microbiol. Infect. 12, 446–452. Oliveira, A., de Cunha, M.L., 2010. Comparison of methods for the detection of biofilm production in coagulase-negative staphylococci. BMC Res. Notes 3, 260-264. Oliveira, A.M., Padovani, C., Miya, N., Sant’Ana, A.S., Pereira, J.L., 2011. Incidence of enterotoxin D producing Staphylococcus spp in Brazilian cow’s raw milk and its relation with coagulase and thermonuclease enzymes. Foodborne Pathog. Dis. 8, 159-163. Omori, G., Kato, Y., 1959. A staphylococcal food-poisoning caused by a coagulase negative strain. Bilken's Journal 2, 92. Otto, M., 2009. Staphylococcus epidermidis–the ‘accidental’ pathogen. Nat. Rev. Microbiol. 7, 555-567. Park, J.Y., Fox, L.K., Seo, K.S., McGuire, M.A., Park, Y.H., Rurangirwa, FR., Sischo, W.M., Bohach, G.A., 2011. Detection of classical and newly described staphylococcal superantigen genes in coagulase-negative staphylococci isolated from bovine intramammary infections. Vet. Microbiol. 147, 149–154. Piechota, M., Kot, B., Frankowska-Maciejewska, A., Woźniak-Kosek, A., 2018. Biofilm Formation by Methicillin-Resistant and Methicillin-Sensitive Staphylococci Strains from Hospitalized Patients in Poland. BioMed. Res. Int. Volume 2018, Article ID 4657396, 7 pages. Pinheiro, L., Brito, C.I., de Oliveira, A., Martins, P.Y.F., Pereira, V.C., daCunha, M.R., 2015. Staphylococcus epidermidis and Staphylococcus haemolyticus: Molecular Detection of Cytotoxin and Enterotoxin Genes. Toxins (Basel) 7, 3688–3699. Pinheiro, L., Brito, C.I., de Oliveira, A., Pereira, V.C., da Cunha, M.L.R.S., 2016. Staphylococcus epidermidis and Staphylococcus haemolyticus: detection of biofilm genes and biofilm formation in blood culture isolates from patients in a Brazilian teaching hospital. Diagn. Microbiol. Infect. Dis. 86,11-14. Podkowik, M., Park, J.Y., Seo, K.S., Bystroń, J., Bania, J., 2013. Enterotoxigenic potential of coagulase-negative staphylococci. Int. J. Food Microbiol. 163, 34–40. Prakash, P.H., Rajan, V., Gopal, S., 2016. Predominance of SCCmec type IV and V among biofilm producing device-associated Staphylococcus aureus strains isolated from tertiary care hospitals in Mysuru, India. Enferm Infecc Microbiol. Clín. 35, 229-235. Prasad, S., Nayak, N., Satpathy, G., Nag, H.L., Venkatesh, P., Ramakrishnan, S., Ghose, Supriyo, Nag, T.C., 2012. Molecular & phenotypic characterization of Staphylococcus epidermidis in implant related infections. Indian J. Med. Res. 136, 483-490. Qin, Z., Yang, X., Yang, L., Jiang, J., Ou, Y., Molin, S., Qu, D., 2007. Formation and properties of in vitro biofilms of ica-negative Staphylococcus epidermidis clinical isolates. J. Med. Microbiol. 56, 83–93. Rall, V.L., Sforcin, J.M., de Deus, M.F., de Sousa, D.C., Camargo, C.H., Godinho, N.C., Galindo, L.A., Soares, T.C., Araújo Júnior, J.P., 2010a. Polymerase chain reaction detection of enterotoxins genes in coagulase-negative Staphylococci isolated from Brazilian Minas cheese. Foodborne Pathog. Dis. 7, 1121-1123. Rall, V.L.M.I., Sforcin, J.M.I., Augustini, V.C.M.I., Watanabe, M.T.I., Fernandes, A.I., Rall, R., Silva, M.G., Araújo, J.P. 2010b. Detection of enterotoxin genes of Staphylococcus sp isolated from nasal cavities and hands of food handlers. Braz. J. Microbiol. 41, 59–65. Rapini, L.S., Cerqueira, M.M., Carmo, L.S., Veras, J.F., Souza, M.R., 2005. Presença de Staphylococcus spp produtores de enterotoxinas e da toxina da síndrome do choque tóxico em manipuladores de queijo de cabra. Arq. Bras. Med. Vet. Zootec 57, 825-829. Rodrigues, M. X., Silva, N.C.C., Trevilin, J.H.T., Cruzado, M.M.B., Mui, T.S., Duarte, F.R.S., Castillo, C.J.C., Canniatti-Brazaca, S.G., Porto, E., 2017. Molecular characterization and antibiotic resistance of Staphylococcus spp. isolated from cheese processing plants. J. Dairy Sci. 100, 5167–5175. Rodríguez, M., Núñez, F., Córdoba, J.J., Bermúdez, E., Asensio, M.A., 1996. Gram-positive, catalase-positive cocci from dry cured Iberian ham and their enterotoxigenic potential. Appl. Environ. Microbiol. 6, 1897-1902. Rohde, H., Kalitzky, M., Kroger, N., Scherpe, S., Horstkotte, M.A., Knobloch, J.K.M., Zander, A.R., Mack, D., 2004. Detection of virulence-associated genes not useful for discriminating between invasive and commensal Staphylococcus epidermidis strains from a bone marrow transplant unit. J. Clin. Microbiol. 42, 5614-5619. Rojas, M.B., Antonelli, C.M., Pereira, E.P.L., Cunha, M.L., 2012. Detection of enterotoxin a in coagulase-negative staphylococci isolated from nutrition students. Archives of Clinical Microbiology 3, 6. Rosec, J.P., Guiraud, J.P., Dalet, C., Richard, N., 1997. Enterotoxin production by Staphylococci isolated from foods in France. Int. J. Food Microbiol. 35, 213-221. Sahal, G., Bilkay, I.S., 2014. Multi drug resistance in strong biofilm forming clinical isolates of Staphylococcus epidermidis. Braz. J. Microbiol. 45, 539-544. Saising, J., Singdam, S., Ongsakul, M., Voravuthikunchai, S.P., 2012. Lipase, protease, and biofilm as the major virulence factors in staphylococci isolated from acne lesions. Biosci. Trends. 6, 160-164. Salgueiro, V.C., Iorio, N.L., Ferreira, M.C., Chamon, R., dos Santos, K.R., 2017. Methicillin resistance and virulence genes in invasive and nasal Staphylococcus epidermidis isolates from neonates. BMC Microbiol. 17, 15–25. Santos, D.A., 2003. O papel do manipulador de alimentos em surtos de intoxicação alimentar causados por espécies de Staphylococcus ocorridos em quatro cidades do Estado de Minas Gerais, Brasil. 85f. Dissertação (Mestrado em Ciências Biológicas) - Instituto de Ciências Biológicas, Universidade Federal de Minas Gerais, Belo Horizonte. Schlegelova, J., Babak, V., Holasova, M., Dendis, M., 2008. The biofilm positive Staphylococcus epidermidis isolates in raw materials, foodstuffs and on contact surfaces in processing plants. Folia Microbiol. 53, 500-504. Schommer, N.N., Christner, M., Hentschke, M., Ruckdeschel, K., Aepfelbacher, M., Rohde, H., 2011. Staphylococcus epidermidis uses distinct mechanisms of biofilm formation to interfere with phagocytosis and activation of mouse macrophage-like cells 774A.1. Infect. Immun. 79, 2267–2276. Siqueira, A. K., Salerno, T., Lara, G.H.B., Condas, L.A.Z., Pereira, V.C., Riboli, D.F.M., LISTONI, F.J.P., Da Silva, A.V., Leite, D.S., Da Cunha, M.L.R.S., Ribeiro, M.G., 2017. Enterotoxin Genes, Multidrug Resistance, And Molecular Typing of Staphylococcus Spp. Isolated from Organic Bovine Milk. Braz. J. Vet. Res. Anim. Sci. 54, 81-87. Smith, K., Ramage, G., Lappin, D., Lang, S., 2008. Biofilm formation by Scottish clinical isolates of Staphylococcus species. J. Med. Microbiol. 57, 1018–1023. Sospedra, I., Marín, R., Mañes, J., Soriano, J.M., 2012. Rapid whole protein quantification of staphylococcal enterotoxin B by liquid chromatography. Food. Chem. 133, 163-166. Spiliopoulou, A.I., Kolonitsiou, F., Krevvata, M.I., Leontsinidis, M., Wilkinson, T.S., Mack, D., 2012. Bacterial adhesion, intracellular survival and cytokine induction upon stimulation of mononuclear cells with planktonic or biofilm phase Staphylococcus epidermidis. FEMS Microbiol. Lett. 330, 56–65. Stepanović, S., Vuković, D., Dakic, I., Savić, B., Vlahović, M.S., 2000. A modified microtiter-plate test for quantification of staphylococcal biofilm formation. Journal of Microbiological Methods 40, 175-179. Udo, E.E., Al-Bustan, M.A., Jacob, L.E., Chugh, T.D., 2009. Enterotoxin production by coagulase negative staphylococci in restaurant workers from Kuwait City may be a potential cause of food poisoning. Journal of Medical Microbiology 48, 819-823. Valle, J., Gomez-Lucia, E., Piriz, S., Goyache, J., Orden, J.A., Vadillo, S., 1990. Enterotoxin production by Staphylococci isolated from healthy goats. Appl. Environ. Microbiol. 56, 1323–1326 Vasconcelos, N.G., Pereira, V.C., Araújo Júnior, J.P., Da Cunha Mde, L., 2011. Molecular detection of enterotoxins E, G, H and I in Staphylococcus aureus and coagulase negative Staphylococci isolated from clinical samples of newborns in Brazil. J. Appl. Microbiol. 111, 749–762. Veras, J.F., Santos, D.A., Carmo, L.S., 2003. Levantamento de surtos de toxinfecção alimentar envolvendo leite e produtos derivados no estado de Minas Gerais, Brasil. Higiene Alimentar 17, 218-222. Vuong, C., Otto, M., 2002. Staphylococcus epidermidis infections. Microbes Infect. 4, 481–489. Weir, D., Jonem, C., Ammerman, L., Dybdahl, K., Tomlinson, S., 2007. Report of a strain of Staphylococcus caprae with the genes for enterotoxin A and enterotoxin-like toxin type P. J. Clin. Microbiol. 45, 3476–3477. Zell, C., Resch, M., Rosenstein, R., Albrecht, T., Hertel, C., Götz, F., 2008. Characterization of toxin production of coagulase-negative Staphylococci isolated from food and starter cultures. Int. J. Food Microbiol. 127, 246–251. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||